2020/12/19 - [공부/R] - [R]for 함수를 이용한 데이터 자동 계산
[R]for 함수를 이용한 데이터 자동 계산
몇일전 빅데이터를 분석하기 위한 코딩을 했었다. 2020/12/15 - [공부/R] - [R]데이터 핸들링 연습(2020.12.15) 물론 엑셀로 작업하는 것보다 훨씬... 빠른 속도로 분석을 하였다. 하지만...? 자동화가 되어
fishingbass.tistory.com
이전 포스팅에서 for 함수를 이용한 데이터 자동 계산에 대한 내용으로
작성했었다.
사실 for 문을 사용하기 이전에도 비교적 편하게
데이터 분석을 했던 코딩이 있다.
library(readxl)
library(dplyr)
ta <- read_xlsx("c:/한강_수질자료.xlsx") #데이터 불러오기
data.frame(ta)
ta[is.na(ta)] = 0 # NA 값을 0으로 변환
taname <- table(ta$측정소명) #측정소 명 추출
#(xx[3,1]) 숫자 바꾸면서 이름 지정하는 방법
x <- taname
xx <- data.frame(x)
la<- as.character(xx[1,1])
la
ex <- filter(.data = ta, 측정소명 == la) #원하는 측정소 추출
su <- summary(ex) #데이터 구조 확인
en <- data.frame(su[3 , ])
colnames(en) <- la
result <- en #첫번째 결과임, 이걸 만들어 줘야 처음에 이어 붙일 수 있음
result
#두번째 분석
la<- as.character(xx[2,1])
la
ex <- filter(.data = ta, 측정소명 == la) #원하는 측정소 추출
su <- summary(ex) #데이터 구조 확인
en <- data.frame(su[3 , ])
colnames(en) <- la
add <- en
total <- cbind(result, add)
total
#반복되게 하는 법..
#엔터만 누르면 되는데... 이것마저도 귀찮다...
#처음에 a 변수에 몇번째 분석인지 입력해줘야 함
la<- as.character(xx[b,1])
ex <- filter(.data = ta, 측정소명 == la)
su <- summary(ex)
en <- data.frame(su[3 , ])
colnames(en) <- la
add <- en
total <- cbind(total, add)
a <- b + 1
b <- as.numeric(a)나름 생각해서 만든 나만의 분석 방법 이었다.
하지만 너무 복잡했고...
결론적으로는 반복이 되긴 하지만 수동으로
분석을 해야만 했다.
그때 생각났던 함수가 for 인데...
처음에는 이해가 잘 안됬지만
고수분들의 자료를 보고 나만의 방법으로 코딩을 해보았다.
이전의 포스팅에서 했던 내용을 자세히 작성해보고자 한다.
전체 코드는 다음과 같다.
library(readxl)
library(dplyr)
data <- read_xls("c:/wq.xls")
data[is.na(data)] = 0
drowname <- table(data$측정소명)
drowname1 <- data.frame(head(drowname, n = 10))
drowname1 = data.frame(drowname1[ , -2])
colnames(drowname1)<-c("측정소")
drowname1
frt1 <- filter(.data = data, 측정소명 == "삼산천")
frt2 <- summary(frt1)
frt3 <- frt2[3,]
frt4 <- data.frame(frt3)
frt4
f <- for (i in 2:10) {
a = drowname1[i , ]
hap1 = filter(.data = data, 측정소명 == a)
hap2 = summary(hap1)
hap3 = hap2[3,]
hap4 = data.frame(hap3)
frt4[,i] <- hap4
}
colnames(frt4) <- drowname1$측정소
head(frt4)크게 4개의 단락으로 구분해서 작성해 보고자 한다.
Chapter 1.
library(readxl) #엑셀자료를 불러 올 수 있는 package
library(dplyr) #데이터 핸들링 최강의 package?실행
> library(readxl)
> library(dplyr)
다음의 패키지를 부착합니다: ‘dplyr’
The following objects are masked from ‘package:stats’:
filter, lag
The following objects are masked from ‘package:base’:
intersect, setdiff, setequal, unionreadxl = R에서 엑셀을 불러올 수 있게 해주는 package
dplyr = R에서 빅데이터를 핸들링하는데 최강이라고 하는데... 공부 더 해야겠다.. ^^;
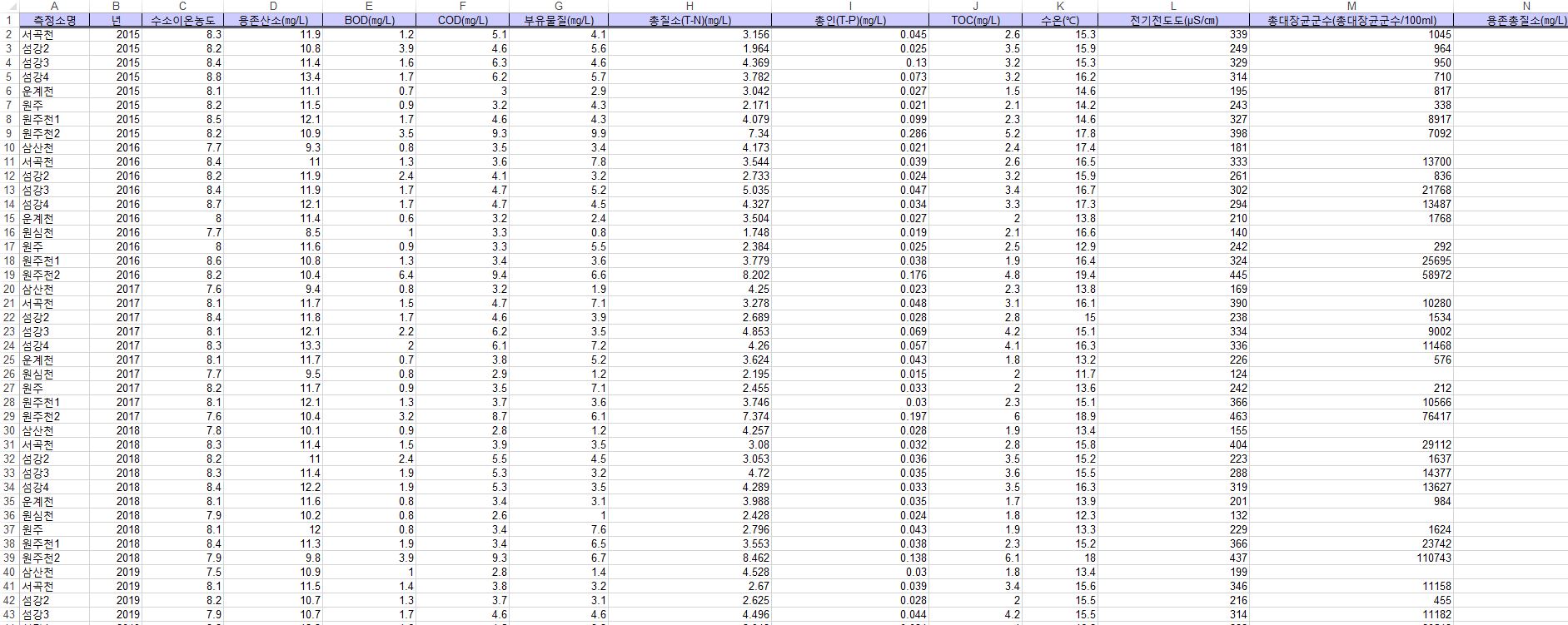
수질측정망의 자료에서 원하는 데이터만 남기고
필요없는 데이터는 살짝 지워준다.
원래 내가 분석하는 데이터는 5천개 이상의 빅데이터 인데...
예시이기 때문에 약 50개 정도의 데이터를 가지고
분석을 해보고자 한다.
Chapter 2.
data <- read_xls("c:/수질_일반측정망.xls") #데이터 불러오기
data[is.na(data)] = 0 #비어있는 cell을 0으로 채워주기
drowname <- table(data$측정소명) #측정소명만 따로 빼기
drowname1 <- data.frame(head(drowname, n = 10))
drowname1 #동일 이름이 몇개 있는지 확인
drowname1 = data.frame(drowname1[ , -2]) #동일 이름 빈도수 제거
colnames(drowname1)<-c("측정소") #열 이름을 측정소로 지정
drowname1 #측정소 이름만 추출
하나 하나 차근차근 살펴보자.
먼저 엑셀로 된 데이터를 불러와 준다.
data <- read_xls("c:/수질_일반측정망.xls") #데이터 불러오기
데이터 중에서 비어있는 곳이 있는데...
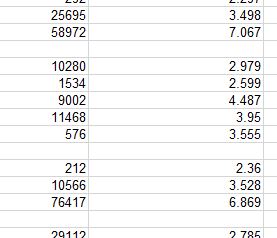
이렇게 비어 있으면 데이터 분석을 할 때 마다
귀찮아지기 때문에
이런부분을 0으로 채워 주기로 하자.
data[is.na(data)] = 0 #비어있는 cell을 0으로 채워주기
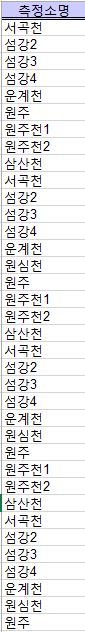
2015~2020년에 수질을 측정한 측정소명이 동일하다.
동일한 측정소 명이 몇개인지 확인이 필요한데...
다음과 같이 확인해 보자.
drowname <- table(data$측정소명) #측정소명만 따로 빼기
drowname1 <- data.frame(head(drowname, n = 10))
drowname1 #동일 이름이 몇개 있는지 확인실행
> drowname <- table(data$측정소명) #측정소명만 따로 빼기
> drowname1 <- data.frame(head(drowname, n = 10))
> drowname1 #동일 이름이 몇개 있는지 확인
Var1 Freq
1 삼산천 5
2 서곡천 6
3 섬강2 6
4 섬강3 6
5 섬강4 6
6 운계천 6
7 원심천 5
8 원주 6
9 원주천1 6
10 원주천2 6동일한 수질측정망 지점의 갯수를 한눈에 확인 할 수 있다.
여기서 내가 필요한 것은 수질측정소 이름이기 때문에
Freq, 즉 빈도수는 제거하도록 하자.
drowname1 = data.frame(drowname1[ , -2]) #동일 이름 빈도수 제거
colnames(drowname1)<-c("측정소") #열 이름을 측정소로 지정
drowname1 #측정소 이름만 추출실행
> drowname1 = data.frame(drowname1[ , -2]) #동일 이름 빈도수 제거
> colnames(drowname1)<-c("측정소") #열 이름을 측정소로 지정
> drowname1 #측정소 이름만 추출
측정소
1 삼산천
2 서곡천
3 섬강2
4 섬강3
5 섬강4
6 운계천
7 원심천
8 원주
9 원주천1
10 원주천2동일한 이름의 측정소 빈도수를 나타내고 있던
2열이 제거된 모습을 확인 할 수 있다.
또한 1열의 열이름이 측정소로 지정되었다.
Chapter 3.
그렇다면 이제 분석하고자 하는 데이터를 먼저 뽑아보자.
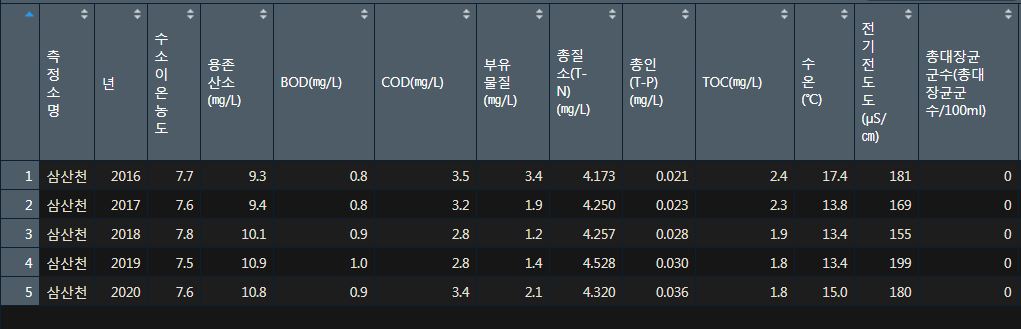
먼저 삼산천 이라고 지정된 수질측정망에서
측정된 데이터의 중앙값을 구해보자.
frt1 <- filter(.data = data, 측정소명 == "삼산천")
frt2 <- summary(frt1)
frt3 <- frt2[3,]
frt4 <- data.frame(frt3)
frt4filter 함수를 이용해서 원래 데이터
즉, 처음에 불러온 자료에서 측정소명이 삼삼천이라고 되어 있는 데이터를
frt에 넣어달라는 명령이다.
다음으로 summary(frt1)을 실행하면...
> frt2 <- summary(frt1)
> frt2
측정소명 년 수소이온농도 용존산소(㎎/L) BOD(㎎/L)
Length:5 Min. :2016 Min. :7.50 Min. : 9.3 Min. :0.80
Class :character 1st Qu.:2017 1st Qu.:7.60 1st Qu.: 9.4 1st Qu.:0.80
Mode :character Median :2018 Median :7.60 Median :10.1 Median :0.90
Mean :2018 Mean :7.64 Mean :10.1 Mean :0.88
3rd Qu.:2019 3rd Qu.:7.70 3rd Qu.:10.8 3rd Qu.:0.90
Max. :2020 Max. :7.80 Max. :10.9 Max. :1.00
COD(㎎/L) 부유물질(㎎/L) 총질소(T-N)(㎎/L) 총인(T-P)(㎎/L) TOC(㎎/L)
Min. :2.80 Min. :1.2 Min. :4.173 Min. :0.0210 Min. :1.80
1st Qu.:2.80 1st Qu.:1.4 1st Qu.:4.250 1st Qu.:0.0230 1st Qu.:1.80
Median :3.20 Median :1.9 Median :4.257 Median :0.0280 Median :1.90
Mean :3.14 Mean :2.0 Mean :4.306 Mean :0.0276 Mean :2.04
3rd Qu.:3.40 3rd Qu.:2.1 3rd Qu.:4.320 3rd Qu.:0.0300 3rd Qu.:2.30
Max. :3.50 Max. :3.4 Max. :4.528 Max. :0.0360 Max. :2.40
수온(℃) 전기전도도(µS/㎝) 총대장균군수(총대장균군수/100ml) 용존총질소(㎎/L)
Min. :13.4 Min. :155.0 Min. :0 Min. :0
1st Qu.:13.4 1st Qu.:169.0 1st Qu.:0 1st Qu.:0
Median :13.8 Median :180.0 Median :0 Median :0
Mean :14.6 Mean :176.8 Mean :0 Mean :0
3rd Qu.:15.0 3rd Qu.:181.0 3rd Qu.:0 3rd Qu.:0
Max. :17.4 Max. :199.0 Max. :0 Max. :0
암모니아성 질소(㎎/L) 질산성 질소(㎎/L) 용존총인(㎎/L) 인산염인(㎎/L) 클로로필 a(㎎/㎥)
Min. :0 Min. :0 Min. :0 Min. :0 Min. :0
1st Qu.:0 1st Qu.:0 1st Qu.:0 1st Qu.:0 1st Qu.:0
Median :0 Median :0 Median :0 Median :0 Median :0
Mean :0 Mean :0 Mean :0 Mean :0 Mean :0
3rd Qu.:0 3rd Qu.:0 3rd Qu.:0 3rd Qu.:0 3rd Qu.:0
Max. :0 Max. :0 Max. :0 Max. :0 Max. :0
분원성대장균군수
Min. :0
1st Qu.:0
Median :0
Mean :0
3rd Qu.:0
Max. :0 다음과 같은 결과를 알 수 있다.
물론 바로 median()을 이용해서 값을 구해도 된다.
여기서 중앙값만 뽑고자...
> frt3 <- frt2[3,]
> frt3
측정소명 년
"Mode :character " "Median :2018 "
수소이온농도 용존산소(㎎/L)
"Median :7.60 " "Median :10.1 "
BOD(㎎/L) COD(㎎/L)
"Median :0.90 " "Median :3.20 "
부유물질(㎎/L) 총질소(T-N)(㎎/L)
"Median :1.9 " "Median :4.257 "
총인(T-P)(㎎/L) TOC(㎎/L)
"Median :0.0280 " "Median :1.90 "
수온(℃) 전기전도도(µS/㎝)
"Median :13.8 " "Median :180.0 "
총대장균군수(총대장균군수/100ml) 용존총질소(㎎/L)
"Median :0 " "Median :0 "
암모니아성 질소(㎎/L) 질산성 질소(㎎/L)
"Median :0 " "Median :0 "
용존총인(㎎/L) 인산염인(㎎/L)
"Median :0 " "Median :0 "
클로로필 a(㎎/㎥) 분원성대장균군수
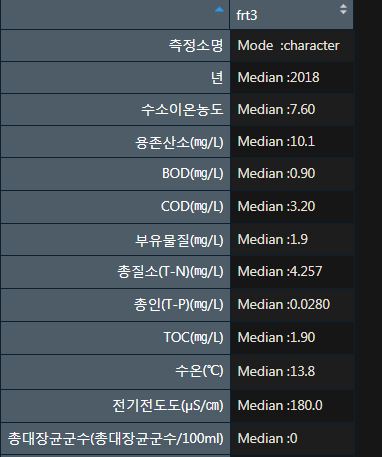
"Median :0 " "Median :0 " 3행의 데이터만 frt3에 넣었다.
frt3을 데이터 프레임 함수를 이용해서
프레임을 만들어 주었다.
> frt4 <- data.frame(frt3)
> frt4
frt3
측정소명 Mode :character
년 Median :2018
수소이온농도 Median :7.60
용존산소(㎎/L) Median :10.1
BOD(㎎/L) Median :0.90
COD(㎎/L) Median :3.20
부유물질(㎎/L) Median :1.9
총질소(T-N)(㎎/L) Median :4.257
총인(T-P)(㎎/L) Median :0.0280
TOC(㎎/L) Median :1.90
수온(℃) Median :13.8
전기전도도(µS/㎝) Median :180.0
총대장균군수(총대장균군수/100ml) Median :0
용존총질소(㎎/L) Median :0
암모니아성 질소(㎎/L) Median :0
질산성 질소(㎎/L) Median :0
용존총인(㎎/L) Median :0
인산염인(㎎/L) Median :0
클로로필 a(㎎/㎥) Median :0
분원성대장균군수 Median :0 2015~2020년
삼산천의 연간 수질자료의
중앙값을 볼 수 있다.
이 방법을
for 함수에 적용해 보았다.
Chapter 4.
이제 for문을 활용해서 나머지 데이터를
한번에 분석해보자.
f <- for (i in 2:10) {
a = drowname1[i , ]
hap1 = filter(.data = data, 측정소명 == a)
hap2 = summary(hap1)
hap3 = hap2[3,]
hap4 = data.frame(hap3)
frt4[,i] <- hap4
}
colnames(frt4) <- drowname1$측정소
head(frt4)f <- for (i in 2:10) {
i 에 2~10의 숫자를 넣어주는 것인데...
왜 2~10의 숫자를 넣어주었냐 하면?!

수질측정망 이름이 10개 이기 때문에 10까지 설정해 준 것이다.
그렇다면 1은 왜 뺐냐?
1은 삼산천 이었기 때문에.. ㅎㅎ
a = drowname1[i , ]
a라는 곳에
측정소 이름이 i 번째에 있는 값을
넣어라
라는 의미를 나타낸다.
hap1 = filter(.data = data, 측정소명 == a)
그래서 hap1에
측정소명이 a랑 같은 이름을 가지고 있는
data를 가지고 와라
hap2 = summary(hap1)
hap3 = hap2[3,]
hap4 = data.frame(hap3)
이거는 chapter 3.과 동일한 내용이라서 pass
그래서 결론적으로
frt4[,i] <- hap4 이다.
frt4의 i열에 데이터를 계속 추가하라는 명령이다.
측 계속 데이터가 2열, 3열, 4열.... 로 추가되는 것
실행을 해보면....?
> f <- for (i in 2:10) {
+ a = drowname1[i , ]
+ hap1 = filter(.data = data, 측정소명 == a)
+ hap2 = summary(hap1)
+ hap3 = hap2[3,]
+ hap4 = data.frame(hap3)
+ frt4[,i] <- hap4
+ }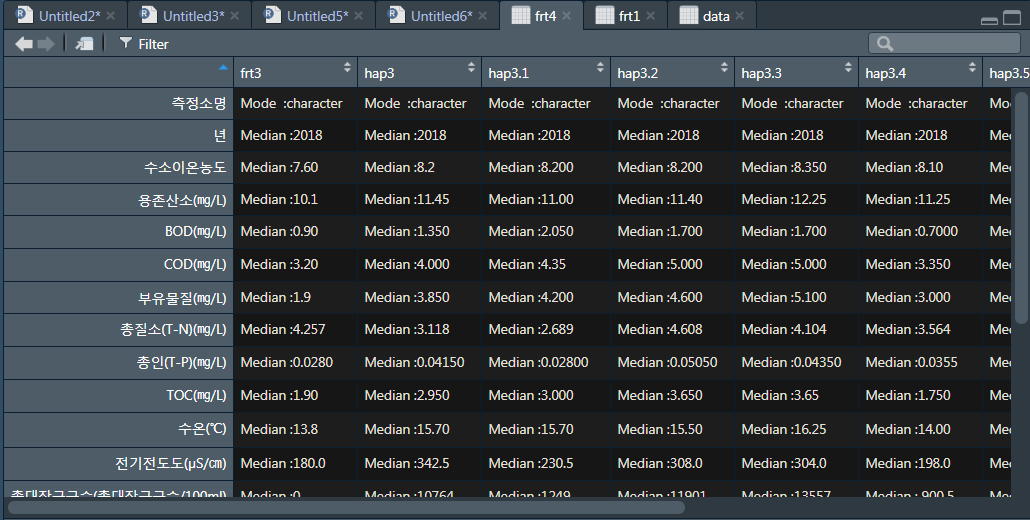
데이터가 잘 들어가 있는 모습을 확인 할 수 있다.
이제 열 이름을 측정소 이름으로 변경해 주면
한눈에 더 쉽게 볼 수 있을 것이다.
colnames(frt4) <- drowname1$측정소
head(frt4)실행
> head(frt4)
삼산천 서곡천 섬강2
측정소명 Mode :character Mode :character Mode :character
년 Median :2018 Median :2018 Median :2018
수소이온농도 Median :7.60 Median :8.2 Median :8.200
용존산소(㎎/L) Median :10.1 Median :11.45 Median :11.00
BOD(㎎/L) Median :0.90 Median :1.350 Median :2.050
COD(㎎/L) Median :3.20 Median :4.000 Median :4.35
섬강3 섬강4 운계천
측정소명 Mode :character Mode :character Mode :character
년 Median :2018 Median :2018 Median :2018
수소이온농도 Median :8.200 Median :8.350 Median :8.10
용존산소(㎎/L) Median :11.40 Median :12.25 Median :11.25
BOD(㎎/L) Median :1.700 Median :1.700 Median :0.7000
COD(㎎/L) Median :5.000 Median :5.000 Median :3.350
원심천 원주 원주천1
측정소명 Mode :character Mode :character Mode :character
년 Median :2018 Median :2018 Median :2018
수소이온농도 Median :7.90 Median :8.200 Median :8.300
용존산소(㎎/L) Median :10.20 Median :11.55 Median :11.45
BOD(㎎/L) Median :0.80 Median :0.9000 Median :1.300
COD(㎎/L) Median :2.9 Median :3.350 Median :3.500
원주천2
측정소명 Mode :character
년 Median :2018
수소이온농도 Median :7.900
용존산소(㎎/L) Median :10.35
BOD(㎎/L) Median :3.700
COD(㎎/L) Median :9.00
> head(frt4)
삼산천 서곡천 섬강2
측정소명 Mode :character Mode :character Mode :character
년 Median :2018 Median :2018 Median :2018
수소이온농도 Median :7.60 Median :8.2 Median :8.200
용존산소(㎎/L) Median :10.1 Median :11.45 Median :11.00
BOD(㎎/L) Median :0.90 Median :1.350 Median :2.050
COD(㎎/L) Median :3.20 Median :4.000 Median :4.35
섬강3 섬강4 운계천
측정소명 Mode :character Mode :character Mode :character
년 Median :2018 Median :2018 Median :2018
수소이온농도 Median :8.200 Median :8.350 Median :8.10
용존산소(㎎/L) Median :11.40 Median :12.25 Median :11.25
BOD(㎎/L) Median :1.700 Median :1.700 Median :0.7000
COD(㎎/L) Median :5.000 Median :5.000 Median :3.350
원심천 원주 원주천1
측정소명 Mode :character Mode :character Mode :character
년 Median :2018 Median :2018 Median :2018
수소이온농도 Median :7.90 Median :8.200 Median :8.300
용존산소(㎎/L) Median :10.20 Median :11.55 Median :11.45
BOD(㎎/L) Median :0.80 Median :0.9000 Median :1.300
COD(㎎/L) Median :2.9 Median :3.350 Median :3.500
원주천2
측정소명 Mode :character
년 Median :2018
수소이온농도 Median :7.900
용존산소(㎎/L) Median :10.35
BOD(㎎/L) Median :3.700
COD(㎎/L) Median :9.00 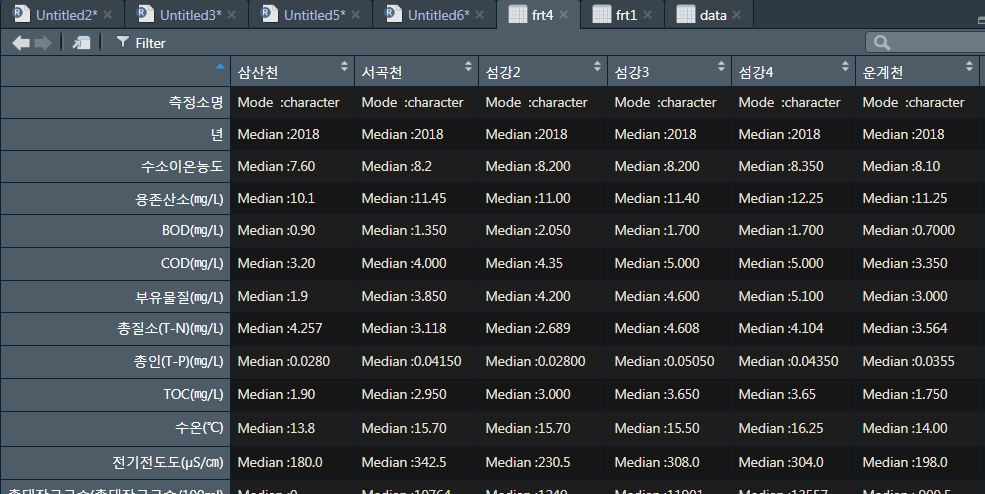
열 이름까지 잘 지정해주었고...
내가 원하는 데이터를 빠르고 쉽게
분석할 수 있게 되었다..
누군가에게는 더 쉽게 할 수 있는데
어렵게 했냐고 생각할 수도 있지만...
누군가에게는 도움이 되길 바라며....
'공부 > R & Python' 카테고리의 다른 글
| [R]R에서 만든 data를 csv 파일로 저장하는 방법 (0) | 2020.12.29 |
|---|---|
| [R]geom_bar, 막대그래프와 데이터 값 표시 (0) | 2020.12.28 |
| [R]for 함수를 이용한 데이터 자동 계산 (0) | 2020.12.19 |
| [R]데이터 핸들링 연습(2020.12.15) (0) | 2020.12.15 |
| [R]r 데이터 프레임 행이름 변경하기, 상관성 분석 시각화 (0) | 2020.11.17 |